Lobnya, Moscow, Russian Federation
Lobnya, Moscow, Russian Federation
A technology has been developed for DNA identification and certification of varieties of meadow clover (Trifolium pratense L.), alfalfa (Medicago varia Mart.), Sowing (M. sativa L.) and hop (M. lupuli-na L.) based on molecular analysis with using SSR and SRAP markers. The recommendations contain a description of the sequence of experiments and protocols for DNA typing procedures. The presented methods were developed by the authors on the basis of their own experimental research and using the data available in the literature. A characteristic of informative primers for each marking system is given, a set of DNA identification markers is proposed, and unique molecular genetic formulas of varieties are drawn up as the basis for a reference genetic passport. Methodological recommendations were prepared with the aim of mastering the technology of DNA certification of forage grasses in practice. Designed for managers and specialists of research and control laboratories, can serve as a textbook for students and postgraduates in specialized specialties.
forage crops, red clover, alfalfa, genetic diversity, DNA-fingerprinting, microsatellite analysis, molecular-genetic certification
1. ТЕРМИНЫ, ОБОЗНАЧЕНИЯ И СОКРАЩЕНИЯ
В настоящих рекомендациях применяются следующие основные термины, определения и сокращения:
Ампликон (амплификат) — ПЦР-продукт, образующийся в процессе реакции.
Амплификация — увеличение числа копий генов (количества ДНК).
ДНК — дезоксирибонуклеиновая кислота.
ДНК-паспортизация — методика оценки сортового материала на основе изучения ДНК-полиморфизма.
ДНК-фингерпринтинг (DNA fingerprinting) — метод определения профиля фрагментов ДНК, основанный на анализе ДНК-полиморфизма.
Маркер молекулярного размера — набор фрагментов ДНК известной длины для определения размера зон, анализируемых в ходе электрофореза.
Микросателлитные (SSR) маркеры — тип ДНК-маркеров, основанных на анализе микросателлитов — простых повторяющихся последовательностей генома длиной 1-6 пар оснований.
ПЦР — полимеразная цепная реакция, в ходе которой происходит синтез целевых фрагментов ДНК и многократное увеличение их концентрации в реакционной смеси до количеств, достаточных для идентификации.
SSR-генотипирование — анализ геномного полиморфизма с использованием SSR-маркеров.
Отжиг (гибридизация) — процесс восстановления нуклеиновой кислоты, при котором одноцепочечные полинуклеотиды образуют двухцепочечную молекулу.
ПААГ — полиакриламидный гель.
п. н. — пары нуклеотидов.
Праймер — короткий фрагмент нуклеиновой кислоты (олигонуклеотид), комплементарный ДНК-мишени; служит затравкой для синтеза комплементарной цепи с помощью фермента ДНК-полимеразы.
ТЕМЕД — тетраметилэтилендиамин.
SDS — додецилсульфат натрия.
EDTA (ЭДТА) — этилендиаминтетрауксусная кислота.
Электрофорез — движение заряженных частиц в растворе под действием электрического поля.
Электрофореграмма — картина, полученная после разделения сложной смеси с помощью электрофореза и специфического проявления.
2. ВВЕДЕНИЕ
Эффективность растениеводства в большой степени обусловлена потенциалом использования современных сортов, который реализуется с учетом факторов сортовой принадлежности и генетической чистоты. В соответствии с задачами «Стратегии научно-технологического развития Российской Федерации» от 2016 года и «Федеральной научно-технической программы развития генетических технологий» на 2019–2027 годы в области агроиндустрии предусмотрено ускоренное внедрение современных методов в селекционные программы. Применение генетических технологий «позволит в короткие сроки создать высокопродуктивные сорта растений с улучшенными пищевыми и технологическими свойствами, устойчивые к неблагоприятным условиям окружающей среды и болезням, что обеспечит повышение эффективности сельскохозяйственного производства и рост конкурентоспособности отечественной продукции на мировых рынках».
Сорта растений относятся к объектам интеллектуальной собственности и охраняются патентами, если являются оригинальными, не имеют аналогов и успешно проходят испытания на отличимость, однородность и стабильность (ООС-тест). Традиционные методы сортовой идентификации на основе морфологических дескрипторов и биохимических признаков значительно уступают современным подходам, основанным на молекулярных ДНК-маркерах, по точности, разрешающей способности и воспроизводимости результатов анализа. Система ДНК-идентификации в настоящее время успешно применяется на практике для ряда культур. Не исключено, что в ближайшее время она будет принята и одобрена Международным союзом по защите новых сортов растений (UPOV) в качестве обязательного элемента тестирования при регистрации нового селекционного достижения.
Молекулярные методы оценки генетического разнообразия, так называемый ДНК-фингерпринтинг, предполагают изучение полиморфизма с разработкой надежного способа записи спектров ДНК, полученных в результате полимеразной цепной реакции (ПЦР). На их основе для каждого сорта можно составить генетический паспорт, который позволит определить уникальность сорта, провести анализ однородности семенного и посадочного материала. Генетическая паспортизация сортов, линий и гибридов может значительно повысить эффективность регистрации и авторской защиты селекционных достижений. Она востребована в семеноводстве — при сертификации и коммерческом распространении семян — и в селекционном процессе — при подборе родительских пар для скрещиваний и выявления генетических маркеров ценных признаков.
Для ДНК-идентификации необходимо предварительное создание эталонных генетических паспортов районированных сортов. Путем сличения с ними тестируемого образца можно установить подлинность сорта, гибридность, наличие примесей и т. д. Внедрение методов ДНК-фингерпринтинга в практику требует комплексного научного подхода, включающего выбор оптимальной системы молекулярного маркирования и создание эффективных технологий генотипирования с учетом особенностей растений конкретного вида.
В Федеральном государственном центре кормопроизводства и агроэкологии имени В. Р. Вильямса разрабатывается система идентификации и паспортизации сортов кормовых культур на основе различных типов ДНК-маркеров. На текущий момент методы определения сортовой принадлежности адаптированы для многолетних бобовых трав; в результате созданы эталонные молекулярно-генетические паспорта для ряда сортов важнейших видов клевера и люцерны.
Разрабатываются генетические паспорта для сортов вики яровой и озимой, рапса, сурепицы и горчицы, некоторых видов злаковых трав селекции ВНИИ кормов.
Методика ДНК-паспортизации сортов кормовых трав на основе различных панелей маркеров включает несколько этапов, которые выполняются в последовательности, представленной схематично на рис. 1.
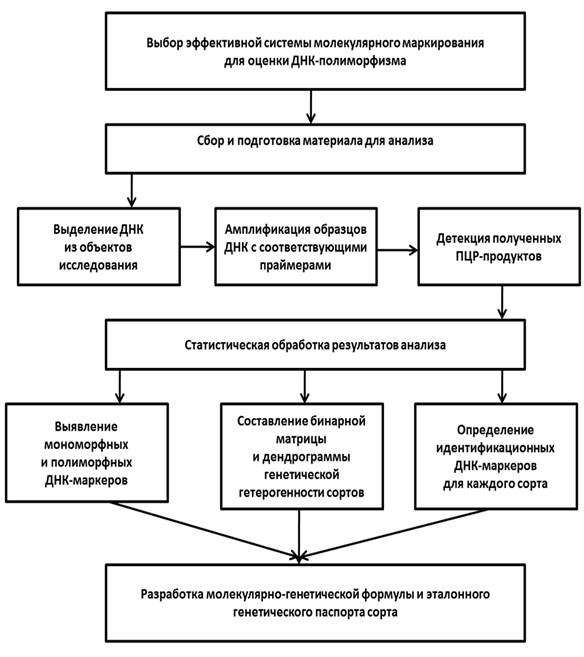
Рис. 1. Основные этапы ДНК-идентификации и генетической паспортизации
сортов кормовых культур
3. ОБОРУДОВАНИЕ И МАТЕРИАЛЫ
Для проведения исследований по изучению ДНК-полиморфизма и генетической паспортизации используется следующее оборудование и материалы:
- Амплификатор «T100 Thermal Cycler» и «iCycler» (Bio-Rad, США). Прибор для проведения ПЦР с функцией температурного градиента.
- ПЦР-бокс LamSystems (Россия). Предназначен для организации изолированного от внешней среды пространства для стерильных работ в ПЦР-лабораториях.
- Спектрофотометрический анализатор нуклеиновых кислот «Nano Spectrophotometer Nabi» (MicroDigital, Корея) для определения концентрации и чистоты выделенных образцов геномной ДНК.
- Электрофорезные камеры «Multi sub maxi Duo» (Cleaver Scientific Ltd, UK), «i-MupidJ» (Япония) для горизонтального агарозного электрофореза; заливочный столик (Хеликон, Россия).
- Электрофорезная камера «Mini-Protean Tetra System» (Bio-Rad, США) для вертикального электрофореза в полиакриламидном геле (ПААГ).
- Универсальный источник питания «Power Pac Basic» (Bio-Rad, США). Предназначен для создания тока определенной силы и напряжения при подключении камер для горизонтального или вертикального электрофореза.
- Система гель-документирования «GelDoc XR Plus» (Bio-Rad, США) в комплекте с пакетом оригинальных программ (Image Lab. Software) используется при детекции нуклеиновых кислот для сохранения и анализа изображений агарозных гелей и ПААГ.
- Мини-центрифуги «Microspin FV-2400» и «Multi-vortexV-32» (BioSan, Латвия). Обеспечивают возможность перемешивания компонентов реакционной смеси при постановке ПЦР и разделения фракций в растворах (ресуспендирование при выделении ДНК).
- Центрифуга «Eppendorf 5804» для разделения жидкостей разных удельных плотностей и отделения жидкости от осадка.
- Твердотельный термостат «TDB-120» (BioSan, Латвия). Необходим для процедур, связанных с нагревом и инкубацией.
- Система получения деионизированной стерильной воды «Milliporo Simplicity UV» (Франция) для приготовления растворов реагентов и реакционной смеси для ПЦР.
- Мини-автоклав (стерилизатор паровой автоматический) ГКа-25-П3 (Россия) для стерилизации растворов и расходных материалов насыщенным паром под давлением.
- Весы лабораторные ВД и ВПТЭ 150-С для точного взвешивания небольших количеств анализируемых образцов или химических веществ.
- Морозильная камера «Philips Whilpool» (Швеция) для хранения образцов ДНК, реактивов, ферментов. Температурный диапазон — от –9 ºС до –20ºС.
- Холодильник комбинированный лабораторный ХЛ-340 «POZIS».
- Настольный измеритель pH «Ohaus Corporation» (США).
- Набор автоматических пипеток-дозаторов переменного объема.
- Одноразовые полипропиленовые микропробирки на 0,2 и 1,5 мл.
- Одноразовые наконечники для микропипеток (10, 200 мкл и 1 мл).
- Штативы для микропробирок и наконечников.
- Микроволновая печь для плавления агарозы.
- Тефлоновые пестики для пробирок на 1,5 мл, либо керамические термостойкие ступки и пестики для гомогенизации растительного материала.
- Стеклянная химическая посуда.
- Халаты и одноразовые резиновые перчатки.
4. МЕТОДИКА ДНК-ФИНГЕРПРИНТИНГА КОРМОВЫХ ТРАВ
С ПОМОЩЬЮ МОЛЕКУЛЯРНЫХ МАРКЕРОВ
НА ОСНОВЕ ПЦР
Принцип метода
Наиболее распространенным и доступным методом выявления генетического полиморфизма и установления филогенетических связей между растениями является метод фрагментного анализа генома с использованием различных маркерных техник на основе полимеразной цепной реакции (ПЦР). ПЦР — это метод амплификации in vitro заданных фрагментов ДНК с полностью или частично известной последовательностью в процессе чередования определенных температурных циклов. Каждый цикл состоит из трех этапов: денатурация — разрыв двухцепочечной структуры ДНК; отжиг — присоединение праймеров к одноцепочечной ДНК-мишени и элонгация, заключающаяся в достраивании и удлинении с помощью Taq-полимеразы второй цепи ДНК с 3'-конца праймера (рис. 2).
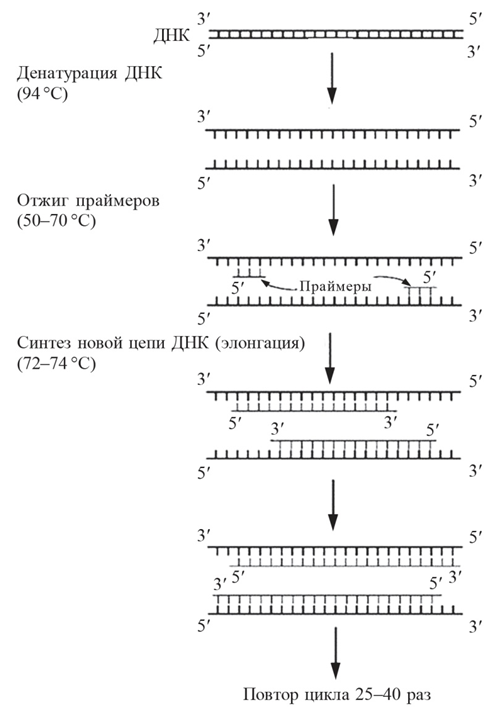
Рис. 2. Полимеразная цепная реакция (https://ru.wikipedia.org/wiki/)
Для проведения ПЦР необходимо наличие в реакционной смеси ряда основных компонентов:
- праймеры — играют ключевую роль в образовании продуктов ампли-фикации, т. к. идентичны соответствующим участкам ДНК-мишени;
- Taq-полимераза — термостабильный фермент, катализирующий полимеризацию ДНК;
- смесь дезоксинуклеотидтрифосфатов (дНТФ) — используется Taq-полимеразой для синтеза второй цепи ДНК;
- буферный раствор (10Х-буфер) — обеспечивает необходимые условия реакции (pH, ионную силу раствора);
- ДНК-матрица — геномная ДНК растения, которую необходимо проанализировать.
- 4. 1. Подготовка материала для анализа
Все работы выполняют с использованием одноразовых расходных материалов, наконечников для автоматических пипеток, пробирок, перчаток и т. д. При отборе материала, а также при подготовке проб для исследования необходимо соблюдать меры, предупреждающие загрязнение образцов объектами внешней среды.
Первым этапом молекулярно-генетического анализа является формирование репрезентативной выборки. Для перекрестноопыляющихся видов, к которым относится большинство кормовых трав, выборка должна включать не менее 30–50 растений от сорта или популяции. Однако масштабный анализ индивидуальных генотипов является сложным, трудоемким и дорогостоящим процессом. Существенно сократить издержки позволяет метод «балк-анализа» (использование суммарной навески части растительной ткани нескольких генотипов в одном образце).
Для ДНК-идентификации клевера лугового и люцерны от каждого сорта случайным образом отбирают по 100 штук семян, стерилизуют в 1,5%-ном растворе перманганата калия (КМnО4) и скарифицируют механическим способом для ускорения прорастания зародыша. Проращивают семена на влажной фильтровальной бумаге в чашках Петри в течение 7–10 дней в зависимости от всхожести конкретного образца. Проростки с семядольными листочками, достигшие размера 1,5 см, отделяют от семенной оболочки и используют для выделения ДНК.
4.2. Выделение ДНК для изучения внутрисортового
и межсортового ДНК-полиморфизма
Экстракция ДНК из растений — исходная точка для молекулярно-генетического анализа. Обычный протокол включает несколько последовательных процедур, обеспечивающих: 1) лизис клеток, для чего растительную ткань гомогенизируют с добавлением лизирующего раствора; 2) освобождение образца от РНК путем внесения в пробирки с образцами фермента РНКазы; 3) осаждение белков и полисахаридов с помощью осаждающего солевого раствора; 4) осаждение ДНК; 5) промывку ДНК в этаноле и растворение в элюирующем растворе (низкосолевой буфер).
Для анализа внутрисортового полиморфизма выделяют ДНК из индивидуальных проростков семян (не менее 10 генотипов от сорта). Для межсортовой дифференциации — из суммарной навески, содержащей не менее 30 проростков. В предварительных экспериментах испытано несколько протоколов ДНК-экстракции. Оптимальным по качеству и выходу ДНК при использовании как единичного проростка, так и общей навески оказался модифицированный протокол на основе SDS-лизирующего буфера, описание которого приводится ниже.
Растворы и реактивы для ДНК-экстракции:
SDS-экстракционный буфер (200 mM трис HCl pH 7,5; 250 mM NaCl; 25 mM ЭДТА; 0,5 % SDS); фермент РНКаза; 5 М ацетат аммония; изопропанол; ЭДТА; этиловый спирт 70 %, охлажденный до –20 ºС;
TE буфер (0,1 М Трис-HCl, 1 мМ ЭДТА, рН 8,0).
Протокол выделения ДНК с использованием
SDS-лизирующего буфера из «балк-образца» проростков семян
- Подготовить для каждого сорта суммарную навеску растительной ткани из 30 проростков семян, отделенных от семенной оболочки;
- Определить объем лизирующего (SDS) буфера на один образец из расчета 300 мкл на 30 мг растительной ткани с учетом общего веса проростков в выборке. Поместить навеску проростков в предварительно промаркированную керамическую ступку;
- Добавить небольшое количество SDS-буфера из объема, рассчитанного на конкретный образец, быстро и энергично растереть ткань в ступке с помощью пестика;
- Продолжить гомогенизацию с оставшимся объемом SDS-буфера до получения однородной суспензии;
- Перенести 300 мкл лизата в 1,5 мл пробирки;
- Добавить в каждую пробирку по 3 мкл РНКазы и перемешать на вортексе;
- Инкубировать смесь 1 час в термостате при температуре 60 ºС;
- Охладить пробирки на льду в течение одной минуты;
- Добавить по 100 мкл 5M ацетата аммония в каждую пробирку, перемешать на вортексе в течение 20 секунд;
- Центрифугировать смесь пять минут при 13000 оборотах. Если образовался недостаточно плотный осадок, повторить центрифугирование;
- Супернатант в полном объеме перенести в чистые 1,5 мл пробирки (не задевая осадок);
- Добавить 300 мкл изопропанола и несколько раз осторожно перемешать переворачиванием;
- Центрифугировать смесь при 13000 оборотах в течение пяти минут;
- Аккуратно слить супернатант и промокнуть пробирки на фильтровальной бумаге;
- Добавить 300 мкл 70%-ного этилового спирта и перемешать несколько раз переворачиванием;
- Центрифугировать две минуты при 13000 оборотах. Осторожно удалить супернатант и промокнуть пробирки на фильтровальной бумаге;
- Открытые пробирки с промытым осадком подсушить при 37 ºС в течение 15 минут до полного испарения спирта;
- Высушенный осадок растворить в 50 мкл ТЕ буфера, перемешать на вортексе и прогреть в термостате при 65 ºС в течение пяти минут.
Протокол выделения ДНК из индивидуальных генотипов сорта для оценки внутрисортового полиморфизма отличается от представленной выше методики по пунктам 1–5. В этом случае в подготовленные полипропиленовые пробирки емкостью 1,5 мл помещают по одному случайным образом выбранному проростку без семенной оболочки (анализируют не менее 10 проростков от каждого сорта). Добавляют 300 мкл экстракционного SDS-буфера (порционно, в два приема), быстро растирают растительную ткань в пробирке с помощью тефлонового пестика и продолжают процедуру в соответствии с пунктами 6–19 основного протокола ДНК-экстракции.
Определение качества и количественного выхода ДНК
Качество и количество выделенной ДНК оценивают путем электрофореза в агарозном геле (1,4%-ный раствор S-агарозы или 1%-ный раствор агарозы типа LE2) и измерением концентрации с помощью спектрофотометра. Если соотношение оптической плотности раствора ДНК (260/280 нм) ниже 1,7–2,0, следует провести дополнительную очистку или повторную ДНК-экстракцию.
4.3. Микросателлитный (SSR) анализ
для молекулярно-генетической паспортизации
Микросателлиты (SSR — simple sequence repeats) — это тандемные повторы простых последовательностей в структуре ДНК. На их основе создают информативные молекулярные маркеры, которые позволяют получить индивидуальную характеристику сорта или генотипа — ДНК-профиль.
Условия проведения ПЦР
Параметры амплификации в значительной степени зависят от ДНК-матрицы, праймеров и типа используемого амплификатора, поэтому условия фрагментного анализа следует оптимизировать для каждой конкретной лаборатории. Состав реакционной смеси для генотипирования на основе SSR-маркеров представлен в таблице 1.
Таблица 1
Состав компонентов реакционной смеси для SSR-анализа
|
Наименование реагента |
На 1 образец (мкл) |
|
H2O (деионизированная) |
9,6 |
|
10x ПЦР-буфер (Taq Turbo Buffer) |
3,0 |
|
50x дНТФ (dNTP mix10 mM each) |
1,0 |
|
100 мкМ праймер (прямой-F) |
0,1 |
|
100 мкМ праймер (обратный-R) |
0,1 |
|
Полимераза (Taq DNA Polymerase) 5 ед./мкл |
0,2 |
|
Образец ДНК 20 нг/мкл |
1,0 |
|
Объем реакционной смеси на 1 пробирку |
15 |
Вносят по 15 мкл реакционной смеси в стерильные пробирки объемом 0,2 мл, тщательно перемешивают и центрифугируют в течение нескольких секунд для удаления пузырьков воздуха. Помещают пробирки с подготовленной реакционной смесью в амплификатор (термоциклер) и задают необходимую программу. Для праймеров к панели SSR-маркеров рекомендуется программа амплификации с высокой начальной температурой отжига для повышения специфичности реакции, а затем снижение температуры через каждые три цикла до оптимальной (+55ºС), так называемая «Touchdown» ПЦР (табл. 2).
Таблица 2
Режим амплификации для ПЦР-анализа с использованием SSR-праймеров
|
Температурные параметры (ºС) |
Время |
Этапы амплификации |
Количество циклов реакции |
||
|
денатурация ДНК |
отжиг праймера |
элонгация |
|||
|
94 |
3 |
|
|
|
1 |
|
94 |
0,5 |
+ |
|
|
3 |
|
68 |
0,5 |
|
+ |
|
|
|
94 |
0,5 |
+ |
|
|
3 |
|
66 |
0,5 |
|
+ |
|
|
|
94 |
0,5 |
+ |
|
|
3 |
|
64 |
0,5 |
|
+ |
|
|
|
94 |
0,5 |
+ |
|
|
3 |
|
62 |
0,5 |
|
+ |
|
|
|
72 |
0,5 |
|
|
+ |
|
|
94 |
0,5 |
+ |
|
|
3 |
|
60 |
0,5 |
|
+ |
|
|
|
72 |
0,5 |
|
|
+ |
|
|
94 |
0,5 |
+ |
|
|
3 |
|
58 |
0,5 |
|
+ |
|
|
|
72 |
0,5 |
|
|
+ |
|
|
94 |
0,5 |
+ |
|
|
30 |
|
55 |
0,5 |
|
+ |
|
|
|
72 |
0,5 |
|
|
+ |
|
|
72 |
10 |
|
|
+ |
1 |
|
4 |
∞ |
||||
По завершении реакции (переход на этап с t +4 ºС) пробирки с анализируемыми образцами можно извлекать из термоциклера и хранить в холодильнике при +4 ºС — кратковременно или сразу использовать для электрофоретического разделения.
Детекция ПЦР-продуктов
Продукты амплификации разделяют методом горизонтального или вертикального электрофореза. Различия в размере ПЦР-продуктов, а следовательно, в скорости перемещения фрагментов ДНК в электрофорезном геле, указывают на разное количество тандемных повторов в исследуемом участке генома.
Предварительный отбор информативных праймеров проводят с помощью горизонтального электрофореза в агарозном геле (1,6%-ный раствор агарозы типа LE2 или 4 % MetaPhorR Agarose). Праймеры, генерирующие отчетливые, воспроизводимые и полиморфные ампликоны, анализируют с применением высокоразрешающего акриламидного геля (10 % ПААГ). Такой электрофорез позволяет обнаружить разницу в длине фрагментов амплификации с точностью до одного нуклеотида и повышает достоверность статистической обработки результатов.
Разделение продуктов ПЦР
методом горизонтального электрофореза в агарозном геле
Растворы и реактивы:
порошок агарозы (1,6 или 4 мг на 100 мл конечного раствора); 1Х ТАЕ — трис-ацетатный электрофорезный буфер (50Х раствор TAE содержит: 2М трис, 1М уксусной кислоты, 0,05М ЭДТА, деионизированную H2O; рН 8,0); 6Х буфер для нанесения образцов на гель; раствор бромистого этидия (0,5 мкг/мл раствора на 100 мл агарозного геля); маркер молекулярного веса.
Протокол горизонтального агарозного электрофореза
- Растворить агарозу в 1Х рабочем растворе ТАЕ;
- Нагревать смесь в микроволновой печи медленно, с интервалами, до полного и равномерного расплавления агарозы;
- Охладить гель до 60 ºС и добавить раствор бромистого этидия;
- В подготовленную кювету с гребенками для создания стартовых лунок залить гель и оставить для застывания;
- После полимеризации агарозы аккуратно извлечь гребенки, стараясь не повредить образовавшиеся лунки, и переместить гель в емкость аппарата для электрофореза, заполненную 1Х ТАЕ;
- Анализируемые продукты ПЦР смешать с загрузочным буфером в соотношении 5 : 1, затем внести в лунки геля с помощью дозатора и одноразовых наконечников;
- Подключить электрофорезную камеру к источнику питания и проводить электрофорез в течение двух часов при 50 V.
Пример: электрофоретическое разделение продуктов ПЦР в агарозном геле (рис. 3).
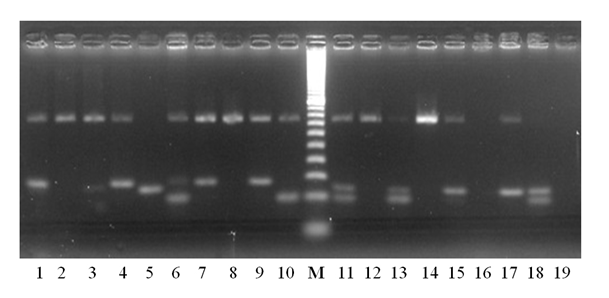
Рис. 3. Электрофореграмма продуктов амплификации образцов
ДНК люцерны с SSR-праймером RCS3711 (4%-ный раствор MetaPhorR Agarose гель). М — маркер молекулярного веса (20 bp DNA Ladder, Takara BIO Inc.). 1–18 — сорта: Луговая 67, Таисия, Селена, Соната, Находка, Мира, Вега 87, Пастбищная 88,
Агния, сортообразец А2, Лада, Вела, Благодать, Сарга, Камелия, Воронежская,
Тамбовчанка, Узень; 19 — контроль (H2O)
Разделение продуктов ПЦР методом вертикального электрофореза в полиакриламидном геле (ПААГ)
Растворы и реактивы:
5Х ТВЕ (трис, борная кислота Na2EDTA*2H2O); 20%-ный водный раствор акриламид (AA)-бисакриламид (19 : 1); трис; ЭДТА; борная кислота хч; 10%-ный персульфат аммония (APS); 50%-ный раствор глицерина водный; бромфеноловый синий; ксиленцианол; H2O деионизированная; раствор бромистого этидия (10 мкл на 200 мл 1X TBE буфера); ТЕМЕD; маркер молекулярного веса.
Протокол вертикального электрофореза
в акриламидном геле (ПААГ)
- Собрать заливочную конструкцию из двух стеклянных пластинок, помещенных в штатив для фиксации, оставив между стеклами зазор для заливки геля;
- Плотно прижать стекла при помощи зажимов на штативе к резиновой прокладке на дне штатива (рис. 4);
- Приготовить 10%-ный раствор ПААГ из 5Х ТВЕ, 20%-ный раствор AA, 50%-ный раствор глицерина, деионизированной H2O. В последнюю очередь добавить в гель TEMED и APS, размешать. Не рекомендуется добавлять TEMED большего объема, чем для двух гелей из-за быстрой полимеризации;
- Оставить на 20 минут для застывания, затем аккуратно извлечь гребенку;
- Промыть лунки геля дистиллированной водой с помощью дозатора;
- Поместить стекла с гелем в модуль, закрепить зажимами;
- Установить модули в электрофорезную камеру (см. рис. 4) и залить 1X TBE-буфером до метки;
- Внести в лунки по 1,5 мкл ПЦР-продукта, смешанного с 1,5 мкл 4Х загрузочного буфера;
- Подключить электрофорезную камеру к источнику питания и проводить электрофорез при следующих условиях: напряжение — 100 V, время — 270 минут, температура +4 ºС.
По окончании электрофореза гель окрашивают в растворе бромистого этидия в течение 15 минут, промывают в дистиллированной воде и фотографируют для сохранения результатов и последующей интерпретации.
Вычисление размера молекул ДНК каждой зоны спектра проводят путем сравнения электрофоретической подвижности данной зоны относительно маркера молекулярного веса. Маркер может представлять собой смесь молекул ДНК с известным размером для каждой фракции. Для SSR-анализа рекомендуется использовать в качестве маркера-стандарта 20 bp DNA Ladder или 100 bp DNA Ladder («Bio-Rad», США). Полиморфными считают фрагменты амплификации, присутствующие на электрофореграммах отдельных сортов или образцов. Анализ и расчет размера фрагментов проводят с помощью специального программного обеспечения.
Рис. 4. Сборка конструкции
для приготовления геля (ПААГ) и рабочих модулей
(Mini-PROTEAN® TetraCell, Instr. Manual, Bio-Rad Laboratories, Inc.)
Пример: электрофоретическое разделение продуктов амплификации ДНК в акриламидном неденатурирующем геле (10 % ПААГ) (рис. 5).
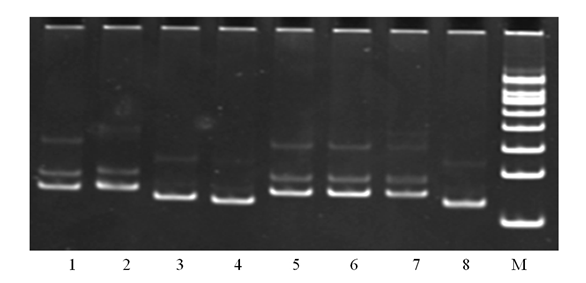
Рис. 5. Электрофореграмма продуктов амплификации плазмид,
содержащих идентификационные последовательности сортов
клевера лугового, с SSR-праймером RCS1307 (10 % ПААГ)
М — маркер молекулярного веса (100 bp DNA Ladder, «Bio-Rad»)
Оценка внутрисортового и межсортового ДНК-полиморфизма
кормовых трав с использованием панели SSR-маркеров
Важным элементом технологии генетической паспортизации является анализ внутрисортового ДНК-полиморфизма. На основании его результатов можно сделать заключение об однородности генотипов сорта и обоснованности использования «балк-образца» для оценки межсортового ДНК-полиморфизма. С этой целью проводят генотипирование 10 индивидуальных проростков семян каждого сорта с использованием трех-пяти наиболее полиморфных микросателлитных маркеров. Если ДНК-спектры индивидуальных образцов идентичны по трем SSR-локусам, можно сделать вывод, что сортовой материал однородный. В этом случае продолжают анализ по остальным маркерам с использованием образца ДНК из общей навески проростков («балк-образец»). Если ДНК-спектры индивидуальных генотипов различаются, сорт гетерогенен. При наличии пяти и большего количества биотипов с различным аллельным составом (анализ частоты встречаемости аллелей), идентификацию или регистрацию сорта (образца) проводить нецелесообразно.
Пример: SSR-анализ внутрисортового ДНК-полиморфизма клевера лугового.
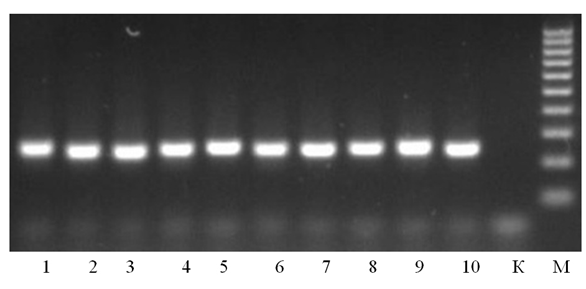
Рис. 6. Продукты амплификации образцов ДНК, выделенных из индивидуальных генотипов сорта Тетраплоидный ВИК, с праймером RCS5600
1–10 — ДНК-пробы генотипов сорта; К — отрицательный контроль (вода);
М — маркер молекулярного веса (100 bp DNA Ladder, «Bio-Rad»)
Отсутствие выраженного полиморфизма между 10 образцами ДНК из отдельных генотипов указывает на генетическую однородность сорта и возможность продолжения SSR-анализа с расширенной выборкой праймеров на «балк-образце», аккумулирующем специфические маркеры, характерные для всей анализируемой популяции.
По результатам тестирования 45 SSR-праймеров к микросателлитным последовательностям генома рекомендованы наборы из восьми маркеров для определения сортовой принадлежности клевера лугового и пяти — для сортов люцерны разных видов (табл. 3, 4). Параметры отбора: 1) максимальный охват групп сцепления гаплоидного набора хромосом изучаемых видов трав; 2) стабильные и воспроизводимые результаты; 3) дискриминационная способность маркера (высокий уровень выявляемого полиморфизма).
Таблица 3
Характеристика микросателлитных (SSR) маркеров для генетической
паспортизации сортов клевера лугового
|
Маркер |
Последовательность |
Мотив |
Группа сцепления |
Диапазон размеров ПЦР-продуктов (п. н.) |
Код |
|
RCS3666 |
F-CATGGCTGCCTGAGGTTAAT |
(AC)20 |
LG6 |
216–230 |
А |
|
R-TCTGTTTCTTGTCTCGGCCT |
|||||
|
RCS3510 |
F-TTCACAAGTTTTTCGGGTGA |
(AT)21 |
LG5 |
249–257 |
B |
|
R-GCCAAAGGGAAGGTTCAATC |
|||||
|
RCS7228 |
F-TCAACAATGTGGCTTCTCCT |
(AT)18 |
LG1 |
179–187 |
C |
|
R-AAGGTTCCCAACCCAATTTC |
|||||
|
RCS4797 |
F-GCCCGTCTACCTTTTGTTCA |
(AT)33 |
LG5 |
155–180 |
D |
|
R-GCGCCATAAGCAACTGTGTA |
|||||
|
RCS2199 |
F-AAAAAGAAAGCGTTAAAGGGG |
(AG)14 |
LG2 |
178–200 |
E |
|
R-GCATTGCCTTTTGCTTCTTC |
|||||
|
RCS5781 |
F-GATCGATCCGAAAACCAAAA |
(AG)16 |
LG3 |
165–210 |
F |
|
R-TGCCATCGAGAGAGAAGGTT |
|||||
|
RCS1307 |
F-CCCTTCTAGCCTAGCAACCA |
(GGGA)22 |
LG4 |
137–158 |
G |
|
R-GCGGAAAAGATTCAGCCTAA |
|||||
|
RCS0017 |
F-TAGTGCTGCTGCACAAATCC |
(AT)16 |
LG7 |
144–177 |
H |
|
R-GGACTTCTCTGATATTGAACTGAATG |
Таблица 4
Характеристика микросателлитных (SSR) маркеров
для генетической паспортизации сортов люцерны разных видов
|
Маркер |
Последовательность нуклеотидов 5'-3' |
Мотив |
Диапазон размеров ПЦР-продуктов (п. н.) |
Код маркера |
|
RCS4982 |
F-TCAACTCTCGTTCCTTCCA |
(AAT)15 |
377–389 |
А |
|
R-ACCAATTGAACTTGAACGCC |
||||
|
RCS4926 |
F-TCAGCAAACAGCTGGTGAAG |
(GGT)17 |
127–145 |
B |
|
R-CTACTCGGTGGGATCGGTTA |
||||
|
RCS3492 |
F-ACTGTCTGGGATGTTGGAGG |
(AC)22 |
174–175 |
C |
|
R-TATCCCTATCATGCGGAACC |
||||
|
RCS3070 |
F-ATGCTTGTGCCCTAACCATC |
(AG)12 |
327–341 |
D |
|
R-TGGTGCTGGAGCTAACATTG |
||||
|
RCS3059 |
F-CTCAATAGGGGGAAGTGCAA |
(AG)17 |
243–255 |
E |
|
R-TGCTCCTCTCTTTGTTGGCT |
Анализ данных
Для определения размеров фрагментов амплификации, полученных с каждым SSR-маркером из набора, используют программы «Gel Analyzer» или «Image Lab» (рис. 7).
а) б)
Рис. 7. Диалоговые окна программ а) «GelAnalyzer» и б) «Image Lab»
Учет проводится по наличию или отсутствию в образце аллелей определенной длины в конкретном локусе. Разные образцы одного и того же сорта должны соответствовать друг другу по длине аллелей.
Составляют бинарную матрицу данных, в которой присутствие фрагмента отмечают как «1», а отсутствие — «0». По матрице данных выбирают праймеры, обеспечивающие стабильную амплификацию и выявляющие полиморфизм фрагментов ДНК изучаемых видов. С помощью «MStools» — надстройки к программе «Excel» — вычисляют показатели генетического разнообразия исследуемого материала: общее количество аллелей на популяцию; частоту встречаемости аллеля; гетерозиготность ожидаемую и наблюдаемую; индекс информативности маркера (PIC). Полученный банк полиморфных ДНК-маркеров можно использовать для молекулярного маркирования хозяйственно ценных признаков и при создании генетического паспорта сорта.
По ДНК-профилю, характерному для каждого сорта, определяют минимальное количество маркеров для его надежной идентификации, буквами латинского алфавита обозначают исследуемые локусы (код локуса), а нижним индексом справа указывают размер выявленных аллелей в парах нуклеотидов. Составленные таким образом молекулярно-генетические формулы служат основой генетического паспорта, где, наряду с информацией по происхождению сорта, регионам возделывания и основным морфобиологическим свойствам, приведены данные по составу аллелей в отдельных локусах генома.
Пример: молекулярно-генетические формулы для сортов клевера лугового и люцерны разных видов с использованием панели SSR-маркеров (табл. 5 и 6).
Таблица 5
Молекулярно-генетические формулы сортов клевера лугового
(Trifolium pratense L.), разработанные на основе SSR-маркеров
|
№ |
Сорт |
Молекулярно-генетическая формула |
|
1 |
Марс |
А216/226B252/252C180/180 D155/169E189/194F192/208G155/155H165/177 |
|
2 |
ВИК 77 |
A219/219B249/249C182/182 D165/174E180180F193/207G156/156H144/144 |
|
3 |
Метеор |
A219/219B254/257C187/187 D170/179E178/189F165/209G142/142H166/172 |
|
4 |
Топаз |
A220/230B252/252C179/179 D157/164E181/190F194/209G151/151H144/150 |
|
5 |
Атлант |
A221/221B252/252C179/184 D158/175E198/200F195/210G137/141H145/145 |
|
6 |
Тетраплоидный ВИК |
A218/230B252/252C179/179 D170/180E181/193F201/205G155/158H144/144 |
Примечание: буквы латинского алфавита — код SSR-маркера; цифровой индекс — размер выявленных аллелей в парах нуклеотидов; жирным шрифтом выделены уникальные аллели для исследуемого локуса.
Таблица 6
Молекулярно-генетические формулы сортов люцерны изменчивой
(Medicago varia Mart.), разработанные на основе SSR-маркеров
|
№ |
Сорт |
Молекулярно-генетическая формула |
|
1 |
Вега 87 |
А377/389B145/127C0D0E0 |
|
2 |
Пастбищная 88 |
А0B0C0D327/341E243/255 |
|
3 |
Благодать |
А0B130/130C174/175D0E0 |
Примечание: жирным шрифтом выделены уникальные аллели для исследуемого локуса; индекс «0» означает нуль-аллель (отсутствие фрагмента по данному локусу).
ДНК-секвенирование для верификации результатов SSR-анализа
ДНК-секвенирование (методы изучения последовательностей фрагментов амплификации, полученных в ПЦР) является наиболее достоверным способом оценки разнообразия, т. к. позволяет обнаружить изменчивость даже на уровне нуклеотидов (полиморфизмы внутри блоков ДНК).
Продукты SSR-генотипирования, используемые для последующей паспортизации, целесообразно отсеквенировать для определения точных размеров выявленных генетических дескрипторов. Секвенирование проводится путем постепенного добавления флуоресцентно-меченных нуклеотидов к целевой ПЦР-матрице. В случае добавления комплементарного нуклеотида детектируется увеличение фонового свечения, что выглядит как пик того или иного цвета (рис. 8).
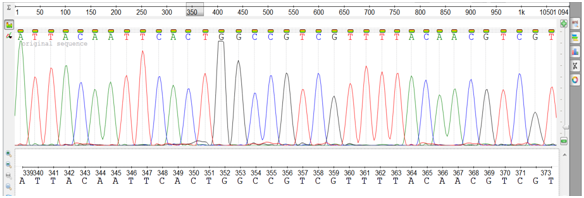
Рис. 8. Хроматограмма, полученная при секвенировании ПЦР-продуктов
на автоматическом анализаторе
С помощью программного обеспечения «OligoCalc» или «Ugene, VectorNTI, AlignX» осуществляют, так называемое «выравнивание» текстового файла с анализируемой последовательностью нуклеотидов относительно последовательности, взятой из базы данных. Анализ данных секвенирования проводится в поисковой системе ВLAST (Basic Local Alignment Search Tool), которая позволяет выявлять как идентичные, так и близкородственные гомологичные последовательности.
Для валидации ДНК-маркеров при разработке генетических паспортов 40 ПЦР-продуктов для клевера лугового и 16 — для люцерны разных видов были клонированы в pAL2-T вектор и секвенированы на автоматическом анализаторе в компании «Евроген» (Россия). Соответствие выявленных фрагментов последовательностям, задепонированным в базе данных, валидизирует результаты анализа и позволяет заключить, что установленные ДНК-маркеры можно использовать в качестве идентификационных для конкретного сорта.
Пример: результаты секвенирования фрагментов амплификации ДНК сортов клевера лугового с SSR-маркером (рис. 9).
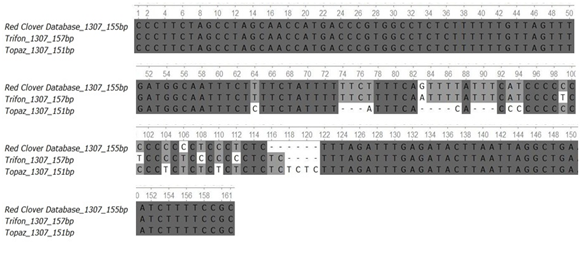
Рис. 9. Сравнительный анализ последовательностей нуклеотидов
SSR-маркера RCS1307 (сорта Трифон, Топаз) и последовательностей этого маркера из базы данных Red Clover Marker Database
С использованием панели SSR-маркеров разработаны молекулярно-генетические паспорта для шести сортов клевера лугового и для трех сортов люцерны изменчивой. Пример паспорта на сорт представлен в приложении 1 и 1а.
-
- Использование панели SRAP-маркеров
для молекулярно-генетической паспортизации
- Использование панели SRAP-маркеров
SRAP-маркирование (sequence-related amplified polymorphism) — относительно новая, простая и надежная техника генотипирования, основанная на использовании в ПЦР пары праймеров, разработанных для амплификации интрон-экзонных участков генома. Определяющую роль в вариабельности продуктов ПЦР играет обратный праймер, нацеленный на некодирующую область генома, обладающую низкой консервативностью. ДНК-полиморфизм, выявленный в этих участках генома с помощью SRAP-маркеров, позволяет установить межсортовые различия у разных видов сельскохозяйственных культур.
На начальном этапе в серии экспериментальных ПЦР отбирают информативные комбинации праймеров для анализа. Отселектированные праймеры (генерирующие воспроизводимые полиморфные ампликоны) используют для анализа всей коллекции образцов. Из 25 испытанных комбинаций праймеров для идентификации сортов люцерны разных видов выбраны семь информативных праймерных комбинаций (табл. 7).
Таблица 7
Характеристика SRAP-маркеров для генетической паспортизации
сортов люцерны
|
Маркерная комбинация |
Последовательность нуклеотидов |
Диапазон размеров ПЦР-продуктов (п. н.) |
Код маркера |
|
F13-Em2 |
CGAATCTTAGCCGGCAC GACTGCGTACGAATTCGG |
125–235 |
А* |
|
F10-R8 |
GTAGCACAAGCCGGAAG GACACCGTACGAATTGAC |
145–708 |
B* |
|
F9-R8 |
GTAGCACAAGCCGGACC GACACCGTACGAATTGAC |
125–385 |
C* |
|
F10-R7 |
GTAGCACAAGCCGGAAG GACTGCGTACGAATTGAG |
165–708 |
D* |
|
F13-R9 |
CGAATCTTAGCCGGCAC GACTGCGTACGAATTTCA |
60–485 |
E |
|
F9-R9 |
GTAGCACAAGCCGGACC GACTGCGTACGAATTTCA |
105–490 |
F |
|
F13-R7 |
CGAATCTTAGCCGGCAC GACTGCGTACGAATTGAG |
50–305 |
G |
*Комбинации праймеров, оказавшиеся информативными для большинства анализируемых сортов и для составления молекулярно-генетических формул.
Условия проведения ПЦР
Реакционная смесь для анализа с использованием SRAP-маркеров аналогична по составу компонентов применяемой при SSR-генотипировании (см. табл. 1), но программа ПЦР отличается по временным и температурным параметрам (табл. 8).
Таблица 8
Режим амплификации для ПЦР-анализа с использованием SRAP-маркеров
|
Температурные параметры |
Время |
Количество циклов ПЦР |
|
94 |
5 |
1 |
|
94 |
1 |
5 |
|
35 |
1 |
|
|
72 |
1 |
|
|
94 |
1 |
35 |
|
50 |
1 |
|
|
72 |
1 |
|
|
4 |
0,02 |
|
|
4 |
∞ |
|
Детекция результатов выполняется с помощью электрофореза в 1,6%-ном агарозном геле (агароза типа LE2, «Lonza», США); для оценки размера полученных фрагментов амплификации используют молекулярный маркер 1kb DNA Ladder («Евроген», Россия).
Анализ данных
Результаты генотипирования анализируют с помощью программного обеспечения «Image Lab» и составляют бинарные матрицы для каждой комбинации праймеров. На основании матриц можно определить индексы генетического сходства и дистанции Нея (Nei, Li, 1979) для попарного сравнения сортов, а затем составить дендрограмму методом UPGMA, если необходимо провести их кластеризацию и установить филогенетические отношения. Показатели генетического разнообразия — процент полиморфизма, эффективное число аллелей на локус, индекс Шеннона (Shannon, Weaver, 1949) — рассчитывают с помощью программы «PopGene».
Пример: Дендрограмма генетической гетерогенности сортов люцерны, выполненная на основе результатов SRAP-анализа (рис. 10).
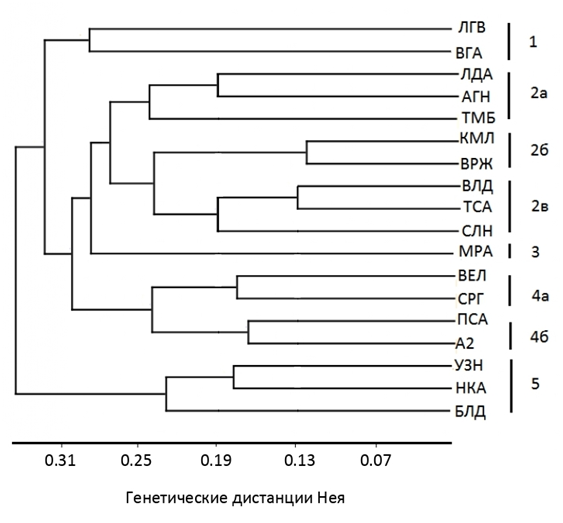
Рис. 10. UPGMA-дендрограмма генетических дистанций
для сортов люцерны разных видов
Примечание: названия сортов обозначены аббревиатурами в виде прописных букв, где ЛГВ-Луговая 67, ВГА-Вега 87, ЛДА-Лада, АГН-Агния, ТМБ-Тамбовчанка, КМЛ-Камелия, ВРЖ-Воронежская 6, ВЛД-Влада, ТСА-Таисия, СЛН-Селена, МРА-Мира, ВЕЛ-Вела, СРГ-Сарга, ПСА-Пастбищная 88, А2-сортообразец А2, УЗН-Узень, НКА-Находка, БЛД-Благодать.
ДНК-спектры исследуемых образцов с информативными комбинациями праймеров можно записать в виде формул и использовать для составления генетического паспорта сорта по панели SRAP-маркеров.
Пример: молекулярно-генетические формулы для сортов люцерны разных видов по панели SRAP-маркеров (табл. 9).
Таблица 9
Молекулярно-генетические формулы сортов люцерны разных видов,
разработанные на основе SRAP-маркеров
|
№ |
Сорт |
Молекулярно-генетическая формула |
|
1 |
Луговая 67 |
A434,358,323,236,185 |
|
2 |
Вега 87 |
A636,317,236,185 |
|
3 |
Вела |
B604,480,410,297,280,193,160,120 |
|
4 |
Узень |
B667,480,410,280,160,120 |
|
5 |
Благодать |
С778,386,354,315,270 |
|
6 |
Селена |
D821,722,605,418 |
|
7 |
Находка |
D808,595,494,435,395 |
|
8 |
Пастбищная 88 |
B704,604,480,410,339,280,208,160,150 C778,394,315,270,202 |
|
9 |
Таисия |
C778,349,315,270,185,163 |
|
10 |
Мира |
С528,425,347,175 A757,625,482,434,228,192,164 |
5. ЗАКЛЮЧЕНИЕ
Разработана система ДНК-идентификации и генетической паспортизации сортов многолетних бобовых трав — клевера лугового (Trifolium pratense L.), люцерны изменчивой (Medicago varia Mart.), посевной (M. sativa L.) и хмелевидной (M. lupulina L.) — по двум панелям молекулярных маркеров (SSR- и SRAP-маркеры). Для ряда сортов составлены уникальные молекулярно-генетические формулы, которые отображают аллельный состав микросателлитных локусов и полиморфизм фрагментов амплификации открытых рамок считывания (интрон-экзонных участков генома). Определены ДНК-идентификационные маркеры для выявления межсортовых различий, верификация которых проведена путем клонирования в pAL2-T вектор и секвенирования по Сэнгеру («Евроген», Россия).
На основе разработанной системы созданы эталонные генетические паспорта для шести районированных сортов клевера лугового и 10 сортов люцерны разных видов. Сличение анализируемого образца с эталонным ДНК-паспортом упростит решение таких задач, как генетическая идентификация, контроль сортовой чистоты и сортового соответствия семенного материала.
Предложенные методы, включая способ выделения ДНК из проростков на основе «балк-стратегии», модифицированные условия ПЦР, детекции и анализа результатов, адаптированы для большинства кормовых культур и являются универсальными для сортовой идентификации и паспортизации.
Приложение 1
Пример: эталонный генетический паспорт клевера лугового
сорт ВИК 77 (разработан на основе панели SSR-маркеров)

Приложение 1а
Структурный элемент эталонного генетического паспорта сорта ВИК 77
(результаты молекулярно-генетического исследования)
Молекулярно-генетическая формула:
A219/219B249/249C182/182D165/174E180/180F193/207G156/156H144/144*
*A – RCS3666; B – RCS3510; C – RCS7228; D – RCS4797; E – RCS2199;
F – RCS5781; G – RCS1307; H – RCS0017.
RCS – Red clover SSR (DOI: 10.1093/dnares/dsi018)
ДНК-идентификационные SSR-маркеры:
|
RCS3510 |
RCS7228 |
|
249 |
182 |
RCS3510 TTC ACA AGT TTT TCG GGT GAA CTT TTG AAA ACA ACT TAT AGT GTG TAT GAA AAC AGC TTA CTT GGT ATT TTT TTT TTT TTG TAG AAA TAG CTA ATA CAA AGG CTT TTA TAT GAT AGT TAA CGT TTA TGC TAT AAG TGC TTA ATT AAG CTA TTT ATC CAC ACA GAA AAG GTG ATA TCA TGG GAT CAT CTG CTG AAT ATC ATA TGC AGC AAA TAA TTA ACA TTC CAG AAA AGA TTG AAC CTT CCC TTT GGC 249
RCS7228 TCA ACA ATG TGG CTT CTC CTA CTT TTT TAC AGT GGC AGG AAA AAT AAT CTT TAG TCA ACA AAC ATT ATC AAA GAA TAT ATA ACC TAC ACT ATT GAT GTT GTA CAA TGA TTC AAC AAT AGC TAT ATA TAT ATT ATG TGC AGT GGT AGA AGC TTA CTC CAA GCT GAA ATT GGG TTG GGA ACC TT 182
(Секвенирование по Сэнгеру, «Евроген», Россия)
ДНК-профиль сорта по результатам электрофореза
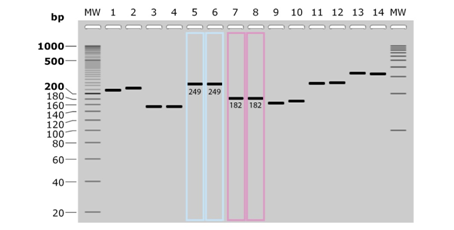
МW — маркер молекулярной массы; 5, 6, 7, 8 — сортоспецифичные ДНК-фрагменты
1. Strategiya nauchno-tehnologicheskogo razvitiya Rossiyskoy Federacii ot 01. 12. 2016 g. № 642. http://www.rcit.su/techinfoM1.html
2. Federal'naya nauchno-tehnicheskaya programma razvitiya geneticheskih tehnologiy na 2019-2027 gody. Postanovlenie ot 22. 04. 2019 g. № 479. http://static.government.ru/media/files/
3. Glik B., Pasternak Dzh. Molekulyarnaya biotehnologiya. Principy i primenenie / Per. s angl. M. : Mir, 2002. 589 s.
4. Kozlov N. N., Klimenko I. A. Tehnologicheskiy pasport kollekcii (regional'naya geneticheskaya kollekciya kormovyh rasteniy). SOP № 5 - «Sozdanie identifikacionnyh DNK-pasportov selekcionnyh dostizheniy kormovyh rasteniy» https://www.vniikormov.ru/pdf/tpk2018.pdf
5. Kutlunina N. A. Molekulyarno-geneticheskie metody v issledovanii rasteniy : ucheb.-metod. posobie; M-vo obrazovaniya i nauki Ros. Federacii, Ural. feder. un-t. Ekaterinburg : Izd-vo Ural. un-ta, 2017. 142 s. ISBN 978-5-7996-2142-1.
6. Osnovy polimeraznoy cepnoy reakcii (PCR). Metodicheskoe posobie / sostavitel' V. V. Zorina. Moskva : OOO «DNK-Tehnologiya», 2012 g. 76 s.
7. Padutov V. E., Baranov O. V., Voropaev E. V. Metody molekulyarno-geneticheskogo analiza. Minsk : Yunipol, 2007. 176 s.
8. Chesnokov Yu. V., Kocherina N. V., Kosolapov V. M. Molekulyarnye markery v populyacionnoy genetike i selekcii kul'turnyh rasteniy. M. : OOO «Ugreshskaya Tipografiya», 2019. 200 s.
9. Don R. H., Cox P. T., Wainwright B. J., Baker K. and Mattick J. S. Touchdown PCR to circumvent spurious priming during gene amplification. Nucleic Acids Res. 1991. 19: 4008.
10. Fu Yong-Bi. Applications of bulking in molecular characterization for plant germplasm: a critical review. Plant Genetic Resources. 2003. 1 (2-3), 161-167. DOI:https://doi.org/10.1079/PGR20032
11. Li G. and Quiros C.F. Sequence-related amplified polymorphism (SRAP), a new marker system based on a simple PCR reaction: its application to mapping and gene tagging in Brassica. Theoretical and Applied Genetics. 2001. V. 103, p. 455-461.
12. Nei M. and Li W. H. 1979. Mathematical Model for Studying Genetic Variation in Terms of Restriction Endonucleases. Proc. Natal Acad. Sci. USA, 76: 5269-5273.
13. Sanger F., Coulson A. R. A rapid method for determining sequences in DNA by primed synthesis with DNA polymerase. J. Mol. Biol. 1975. Vol. 94, p. 444-448.
14. Sato S., Isobe S., Asamizu E., Ohmido N., et al. Comprehensive structural analysis of the genome of red clover (Trifolium pratense L.). DNA Research. 2005. 12(5): 301-364. DOI:https://doi.org/10.1093/dnares/dsi018







